上海金畔生物科技有限公司代理AAT Bioquest荧光染料全线产品,欢迎访问AAT Bioquest荧光染料官网了解更多信息。
MycoLight 比率细菌膜电位试剂盒 红色/绿色荧光
 |
货号 | 22401 | 存储条件 | 在零下15度以下保存, 避免光照 |
| 规格 | 200 Tests | 价格 | 3924 | |
| Ex (nm) | 483 | Em (nm) | 501 | |
| 分子量 | 溶剂 | |||
| 产品详细介绍 | ||||
简要概述
MycoLight 比率细菌膜电位试剂盒是美国AAT Bioquest生产的用于检测细菌的试剂盒,AAT Bioquest的MycoLight 比率细菌膜电位试剂盒使用荧光传感器,在低浓度的革兰氏阳性和阴性细菌细胞中均呈现绿色荧光,但由于较大的染料分子聚集,荧光在较高的细胞溶质浓度下向红色发射转移膜电位。 膜电位的大小随着不同的细菌种类而变化。 对于许多革兰氏阳性物种,红/绿比率倾向于随质子梯度的强度而变化,而在许多革兰氏阴性细菌中,染料的响应似乎与质子梯度强度不成比例。 该试剂盒设计用于在细菌浓度范围为每毫升105-107个生物体时测定细菌膜电位。 可以在510-530nm(FITC滤波片组)和600-660nm(德克萨斯红色滤波片组)下荧光测量染色细胞,激发波长为最常见的激发光源488nm。金畔生物是AAT Bioquest的中国代理商,为您提供最优质的MycoLight 比率细菌膜电位试剂盒。
点击查看光谱
适用仪器
| 流式细胞仪 | |
| 激发: | 488nm激光 |
| 发射: | 530/30nm,610/20nm滤波片 |
| 推荐孔板: | FITC,PE-Texas Red通道 |
| 荧光显微镜 | |
| 激发: | 510/600nm |
| 发射: | 530/660nm |
| 推荐孔板: | 黑色透明 |
| 通道: | FITC/Texas Red滤波片 |
产品说明书
操作方法
1.在任何适当的培养基中培养细菌从对数期培养物中获得健康细菌的最佳结果。在PBS(组分C)或等效的无菌缓冲液中将细菌培养物稀释至~106个细胞/ mL。可以直接从培养基中稀释细菌而不洗涤。准备足够的悬浮液,每次测试提供500 mL。
2.将500μl细菌悬浮液等分到流式细胞仪管中,进行每个染色实验。准备两个额外的管用于去极化控制和未染色的对照。
3.向去极化的对照样品中加入10μl500μMCCCP(组分B)并混合。
4.向每个流式细胞仪管中加入5μlMycoStainIt Green(100X)(组分A)并混合(不要在未染色的对照样品上加入染色剂)。在室温下孵育样品30分钟。可在5分钟后分析染色样品,但信号强度继续增加直至约30分钟。
5.可以在配备有发射488nm的激光的流式细胞仪中测定染色细菌。在绿色(荧光素滤光片)和红色(德克萨斯红色滤光片)通道中收集荧光。应使用对数信号放大收集前向散射,侧向散射和荧光。
6.仪器调整对于检测细菌等相对较小的颗粒尤其重要。使用未染色的对照样品定位前向和侧向散射通道中的细菌群。使用侧向散射作为设置采集触发器的参数。
7.如上所述,在调节流式细胞仪后应用去极化的对照样品。使用正向散射和侧向散射对细菌进行门控并调整荧光光电倍增管电压,使得绿色和红色MFI值近似相等。不要设置补偿。
8.虽然红色和绿色荧光强度的相对量将随着细胞大小和聚集而变化,但红色与绿色荧光强度的比率可以用作膜电位的尺寸无关指示。还可以通过使用正向散射与侧向散射对细菌进行门控来处理数据,并使用红色与绿色荧光的点图分析门控群体,将MFI值报告为线性值,而不是通道。
9.在比率直方图上,在感兴趣的峰周围设置标记并记录平均比值。对于红色与绿色荧光的点图,设置感兴趣群体周围的区域并记录每个的红色和绿色平均荧光强度(MFI)值。为了评估数据,将红色种群MFI除以绿色种群MFI。
10.在流式细胞仪中,细菌仅根据其大小和染色能力进行鉴定。最好通过荧光显微镜检查每个样品,以确认检测到的颗粒确实是细菌。
数据分析
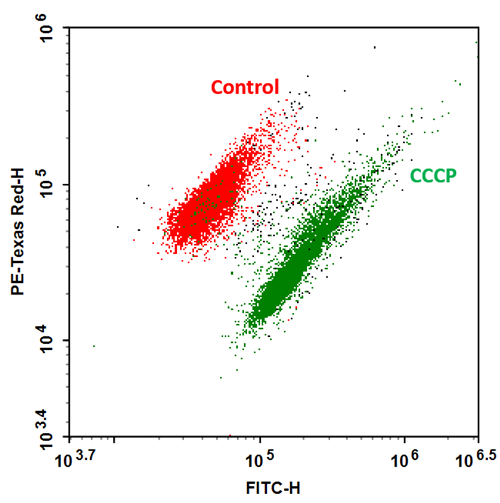
图1.将枯草芽孢杆菌培养至对数期并在PBS中稀释至1×10 6个细胞/ mL的浓度。 然后将细胞用5μMCCCP处理20分钟,并与1X MycoStainIt Green孵育30分钟,然后进行流式细胞术分析。
参考文献
Raman spectroscopic analysis of Lactobacillus rhamnosus GG in response to dehydration reveals DNA conformation changes
Authors: Myintzu Hlaing, M.; Wood, B.; McNaughton, D.; Ying, D.; Augustin, M. A.
Journal: J Biophotonics (2017): 589-597
Antibacterial and antigelatinolytic effects of Satureja hortensis L. essential oil on epithelial cells exposed to Fusobacterium nucleatum
Authors: Zeidan-Chulia, F.; Keskin, M.; Kononen, E.; Uitto, V. J.; Soderling, E.; Moreira, J. C.; Gursoy, U. K.
Journal: J Med Food (2015): 503-6
Inactivation of Cronobacter sakazakii in reconstituted infant formula by combination of thymoquinone and mild heat
Authors: Shi, C.; Jia, Z.; Chen, Y.; Yang, M.; Liu, X.; Sun, Y.; Zheng, Z.; Zhang, X.; Song, K.; Cui, L.; Baloch, A. B.; Xia, X.
Journal: J Appl Microbiol (2015): 1700-6
Fourier transform infra-red spectroscopy and flow cytometric assessment of the antibacterial mechanism of action of aqueous extract of garlic (Allium sativum) against selected probiotic Bifidobacterium strains
Authors: Booyens, J.; Thantsha, M. S.
Journal: BMC Complement Altern Med (2014): 289
Deposition and survival of Escherichia coli O157:H7 on clay minerals in a parallel plate flow system
Authors: Cai, P.; Huang, Q.; Walker, S. L.
Journal: Environ Sci Technol (2013): 1896-903
Observation of injured E. coli population resulting from the application of high-pressure throttling treatments
Authors: De Lamo-Castellvi, S.; Toledo, R.; Frank, J. F.
Journal: J Food Sci (2013): M582-6
Effect of air drying on bacterial viability: A multiparameter viability assessment
Authors: Nocker, A.; Fernandez, P. S.; Montijn, R.; Schuren, F.
Journal: J Microbiol Methods (2012): 86-95
patients and environment
Authors: Lindback, T.; Rottenberg, M. E.; Roche, S. M.; Rorvik, L. M., The ability to enter into an avirulent viable but non-culturable (VBNC) form is widespread among Listeria monocytogenes isolates from salmon
Journal: Vet Res (2010): 8
Behaviors of physiologically active bacteria in water environment and chlorine disinfection
Authors: Sawaya, K.; Kaneko, N.; Fukushi, K.; Yaguchi, J.
Journal: Water Sci Technol (2008): 1343-8
Long-term survival of Legionella pneumophila in the viable but nonculturable state after monochloramine treatment
Authors: Alleron, L.; Merlet, N.; Lacombe, C.; Frere, J.
Journal: Curr Microbiol (2008): 497-502
